Il ciliegio dolce (Prunus avium L.) è una delle specie frutticole perenni più importanti dal punto di vista economico che crescono nelle regioni temperate. Tuttavia, in parte a causa dei cambiamenti climatici e delle pressioni biologiche esercitate dai patogeni, la produzione di ciliegie dolci in Francia è in declino dagli anni Ottanta. Nel frattempo, la selezione tradizionale è un processo lungo a causa della prolungata fase giovanile e della complessità dei caratteri poligenici.
I progressi della ricerca genetica, tra cui la mappatura dei locus quantitative (QTL) e gli studi di associazione genome-wide (GWAS), hanno identificato i marcatori per i tratti chiave, ma sono limitati dalla necessità di un'ampia genotipizzazione. La genotipizzazione per sequenziamento (GBS) emerge come una soluzione promettente, in quanto offre un metodo economico per l'analisi di SNP ad alta densità per migliorare le strategie di selezione.
Nel settembre 2023, Horticulture Research ha pubblicato una ricerca intitolata "Genome-wide association mapping in a sweet cherry germplasm collection (Prunus avium L.) reveals candidate genes for fruit quality traits".
Questo studio ha dapprima utilizzato una collezione di germoplasma di 116 accessioni di ciliegio dolce per analizzare 23 tratti agronomici di qualità del frutto nell'arco di 2-6 anni, osservando un'elevata variazione fenotipica dei tratti di qualità del frutto.
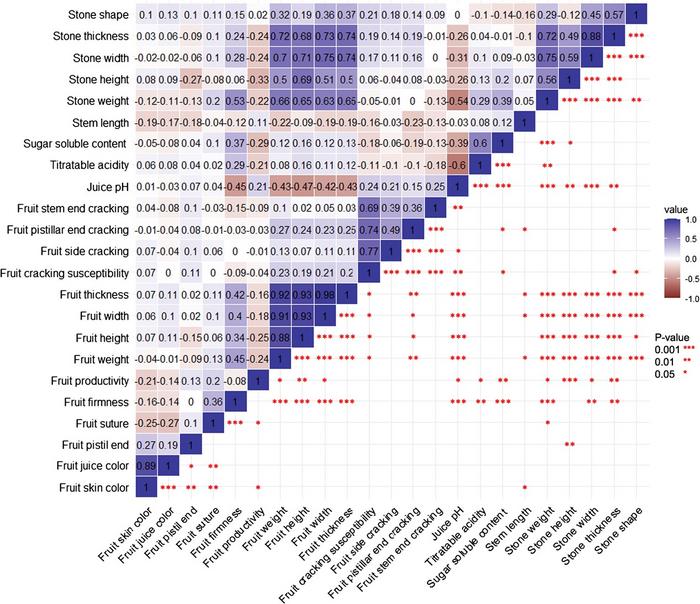 Immagine 1: Matrice di correlazione di Pearson dei 23 tratti studiati.
Immagine 1: Matrice di correlazione di Pearson dei 23 tratti studiati.
Successivamente, sono stati analizzati gli SNP mediante un approccio di genotipizzazione per sequenziamento (GBS).Sono stati condotti studi di associazione genomica (GWAS) con 28 198, 34 864 e 33 760 SNP nei genomi di P. avium var. 'Regina', P. avium var. 'Satonishiki' e Prunus persica 'PLov22n', rispettivamente.Inoltre, attraverso il GWAS, utilizzando due modelli multilocali e tre genomi di riferimento, sono state identificate 65 associazioni SNP-trait uniche per otto tratti.
I ricercatori hanno anche individuato diversi geni candidati coinvolti nei metabolismi dei fitormoni, del calcio e della parete cellulare.
Al fine di identificare associazioni SNP-trait potenzialmente colocalizzanti per i tratti qualitativi della frutta, i ricercatori hanno riassunto i risultati di questo studio e di nove articoli pubblicati in precedenza, e sono stati identificati 11 e 12 geni candidati correlati rispettivamente alle dimensioni dei frutti (peso, larghezza e spessore dei frutti) e ai tratti legati alla spaccatura dei frutti.
La robusta metodologia dello studio ha compreso la caratterizzazione della variazione fenotipica, rivelando una significativa variabilità tra i tratti esaminati, con un'elevata ereditabilità ad ampio spettro per molti di essi, che suggerisce una forte componente genetica.
Il sequenziamento GBS ha prodotto un ricco set di SNP che, dopo il filtraggio della qualità e della frequenza degli alleli minori, ha facilitato l'identificazione di associazioni genetiche tra più caratteri. L'analisi della struttura della popolazione ha rivelato sottopopolazioni distinte, contribuendo a una più profonda comprensione della diversità genetica all'interno della collezione.
Le associazioni SNP-tratto identificate, supportate da un'accurata revisione della letteratura per il confronto e le potenziali colocalizzazioni delle associazioni marcatore-tratto, migliorano la nostra comprensione del controllo genetico della qualità dei frutti nel ciliegio dolce.
Nel complesso, queste conoscenze sono alla base dello sviluppo di strategie di selezione assistita da marcatori, volte ad accelerare gli sforzi di selezione per soddisfare sia le preferenze dei consumatori sia le esigenze dei produttori, affrontando così la complessità dell'architettura genetica del ciliegio dolce e il suo impatto sui tratti qualitativi del frutto.
Fonte: EurekAlert!
Immagini: Horticulture Research
Cherry Times - Tutti i diritti riservati











